牛津团队推出百万级数据抗体-抗原模型,超大模型的上限到底在哪里?
- 2025-07-22 11:55:00
将 ScienceAI 设为星标
第一时间掌握
新鲜的 AI for Science 资讯


编辑丨%
抗体药物是抗癌、抗病毒的「利器」,但其疗效好坏,核心看抗体与抗原的结合强度(ΔΔG)。长期以来,这个关键指标的预测难住了无数科研人员 —— 要么靠昂贵的实验测量,要么依赖 AI 模型却因数据不足屡屡翻车。
近日,牛津大学的研究团队开发了 Graphinity,一种直接从抗体-抗原结构构建的等变图神经网络架构,虽然在 ΔΔG 预测上的测试皮尔逊相关系数(Pearson Correlation Coefficient)——r可达 0.87,但也同样陷入了过拟合的困境。
故而,他们构建接近 100 万个 FoldX 生成与超过两万个合成数据集,以研究预测 ΔΔG 所需的数据量和类型。
他们的研究以「Investigating the volume and diversity of data needed for generalizable antibody–antigen ΔΔG prediction」为题,于 2025 年 7 月 8 日刊登在《Nature Computational Science》

论文链接:https://www.nature.com/articles/s43588-025-00823-8
为何抗体开发困难重重
抗体通过特异性结合靶抗原来介导其生理和治疗功能。因此,在确定和优化先导候选物时,控制亲和力是主要考虑因素,但是传统亲和力定量实验慢得让人抓狂,于是大家只能将希望那个寄托在 ML 上。
FoldX、Rosetta Flex ddG 靠物理方程+经验参数,跑一个突变几分钟到几小时,精度随缘。
早期 ML 在 AB-Bind 645 突变或 SKEMPI 608 突变上「看似封神」,实则是「见过这道题」。一旦按抗体/抗原序列相似度严格切分,r 直接掉到 0.17–0.26,比瞎猜好不了多少。
团队推出的 Graphinity 将野生型(WT)和突变抗体–抗原复合物的结构作为输入,通过 Siamese EGNN 处理相应的图表示,并预测 ΔΔG。

图 1:Graphinity 架构和合成数据集准备。(图源:论文)
实验的数据集包含来自 29 个复合物的 645 个单点突变,并且实验模型在 10 折交叉验证中达到了惊人的 r= 0.87 。但团队提出,这只是过拟合的结果,而不是真正的学习。
当他们决定把互补决定区(CDR)序列同源性截止值设为 100%长度匹配,r 平均下降了 63%。

图 2:Graphinity 模型在ΔΔG 预测中的性能。(图源:论文)
症结总结到最后其实就是一句话:实验数据太少、太偏。于是实验研究者们决定——开闸防水!先造它个一百万数据,看看 ML 到底需要多大的胃口才能吃饱。
合成超大数据集
团队通过使用 FoldX 对结构抗体数据库(SAbDab)中结构解析复合物的界面进行穷尽突变,生成了近 100 万 ΔΔG 数据点。虽然说 FoldX 使用物理方程和经验测量来生成结合亲和力的预测,并不完全真实,但其捕捉了分子相互作用的关键特征。
在此基础上,Graphinity 在 10 折交叉验证中实现了 r=0.89(90% CDR 序列相似度划分)。这已经取得了显著的成果,那么接下来就是量化准确预测实验值所需的数据量。
测试皮尔逊相关系数 r 仅在使用至少 90,000 个突变进行训练的模型中才开始趋于平稳,达到 0.85,这还是在总计投放了 94,126 个数据集的情况下。
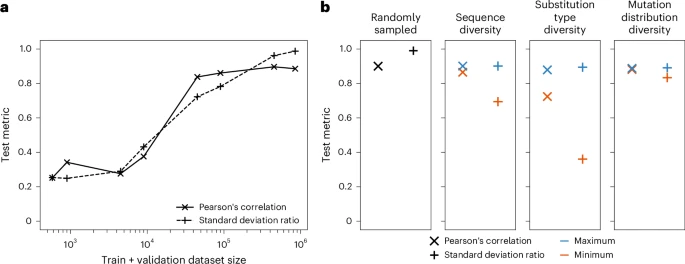
图 3:关于实验ΔΔG 数据集生成的考虑,以提高机器学习预测性。(图源:论文)
在比较预测值和真实值的分布后,团队发现从较小的数据集构建的模型往往会向均值回归,并且尽管预测值没有覆盖真实值的全部范围,但仍然实现了较高的相关性。
那么数据集光有数量还不够,还得有质量——也就是多样性。团队用 10 万个突变子集作为数据源,就三个指标评估数据集的多样性:
序列多样性:从 1177 个抗体降到 75 个,标准差比直接掉 23%;
氨基酸替换类型多样性:把 380 种氨基酸替换压缩到 19 种常见,标准差比再掉 60%;
界面突变结构分布:只让突变集中在界面核心区或外周区,结果……基本没差。
换句话说,抗体序列和替换化学空间的丰富度才是关键,而「在哪儿突变」倒没那么敏感。
最后在 36,391 个实验数据的实战测试里,Graphinity 真切地展示出了它确实能吃透实验分布,而非简单背题:ROC AUC 达到 0.90,平均精度(AP)为 0.82。
数据荒还是存在的
在如此大数量级的实验验证里,研究团队也才将将避开了过拟合的干扰。多次重复试验后,他们得出了一个结论:实验性 ΔΔG 预测的主要挑战在于数据可用性而非模型架构。
他们认为,目前可用的实验数据远远不足,需要更多的数据集,从数十万到数百万不等。本次测试的条件下,团队预估至少需要 90000 个 ΔΔG 值,以实现测试皮尔逊相关系数超过 0.85。
这并非是单纯的重复堆积,而是更加多样的数据类型。目前的实验里在抗体序列同源与氨基酸替换类型方面数据还是很有限的。
故而,团队强调,未来在类似的实验中,需要向更多「机器学习级别的数据」过渡,并寻找更多方法推进通用亲和力预测。
人工智能 × [ 生物 神经科学 数学 物理 化学 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。
 扫码添加微信
扫码添加微信
- 点赞 0
-
分享
微信扫一扫
-
加入群聊
 扫码加入群聊
扫码加入群聊









