谷歌AlphaFold得了诺奖,但DeepMind根本没引用前人论文?
- 2025-07-20 12:49:06

新智元报道
新智元报道
【新智元导读】AlphaFold夺诺奖引争议!2016年,一位博士生在NeurIPS提出的研究,或许正是AlphaFold的「原型」。如今,导师Daniel Cremers发声,质问为何DeepMind忽略这项研究、不加以引用?
AlphaFold因获得诺奖而声名大噪。
在大多数情况下,AlphaFold 2的预测精度几乎可与X射线晶体学相媲美,令人震撼。
生物化学界持续了半个世纪的难题,终于被攻克。
但2016年,Vladimir Golkov博士在NeurIPS大会上就提出,利用深度神经网络从共进化数据中直接预测蛋白质接触图。
在CASP 11测试中,这套方法优于当时其他的所有方法,堪称AlphaFold的「原型」。
最近,慕尼黑机器学习中心主任、慕尼黑工业大学教授Daniel Cremers表示他们团队为AlphaFold获得诺贝尔奖奠定了基础。

如今,Daniel Cremers发问:为何历史上这块奠基石被忽视了?
我们来一探究竟。

在2018年12月,在第13届蛋白质结构预测关键评估CASP(Critical Assessment of protein Structure Prediction)中,AlphaFold 1惊艳亮相,排名第一。
2020年11月,AlphaFold 2在CASP上,大放异彩,中位分数为92.4,接近满分100分;2024年5月8日,AlphaFold 3发布。
但早在2016年AI顶会NeurIPS上,Vladimir Golkov就蛋白质预测作了全会报告。

当时提出的方法论包括:
针对目标氨基酸序列,调用已知三维结构的序列数据库
采用隐马尔可夫模型(HMM)进行多序列比对以识别同源序列
计算突变对共进化统计量
训练深度神经网络直接从原始共进化数据预测蛋白质接触图
在CASP11数据集上的系统评估表明,该方法在精度和速度上均显著超越当时最优技术
这项研究汇聚了深度学习与蛋白质预测领域的多位先驱,包括Thomas Brox、Alexey Dosovitskiy和Jens Meiler等合作者。

论文链接:https://papers.nips.cc/paper_files/paper/2016/file/2cad8fa47bbef282badbb8de5374b894-Paper.pdf
值得玩味的是,在报告结尾,Vladimir已预见性地指出:「架构优化与Scaling将进一步提升性能」——
这与AlphaFold团队的后续突破不谋而合。
至于未被引用的原因,至今尚无定论。
可观看Vladimir当年20分钟的报告,了解更多蛋白质预测的完整发展脉络:
2024年, Demis Hassabis(下图左)和John Jumper(下图右)因蛋白质结构预测的贡献,荣获诺贝尔化学奖。

诺贝尔奖委员介绍了AlphaFold 2的工作原理,大致如下:
序列比对:系统从数据库中寻找与输入序列相似的蛋白质,这些序列可能来自不同物种。通过比对,程序揭示了氨基酸之间的潜在联系。例如,当某个位置突变时,可能与另一个位置的变化相关。
距离图生成:基于序列比对中的相关性信息,程序生成一个距离图,显示氨基酸在空间中的相对距离。
三维结构预测:程序将距离图转换为三维结构,最终以高精度预测蛋白质的形状。

AlphaFold 2的工作原理示意图
Daniel Cremers认为所谓AlphaFold的核心技术思路,其实早已完整呈现在他们2016年的论文中。
他感觉诺贝尔奖委员会可能遗漏了他们的奠基性工作。
对此,AlphaFold 1核心团队成员Hugo Penedone提供了AlphaFold诞生初期的一些历史细节。

AlphaFold 1初始团队的成员Hugo Penedone则还原了DeepMind的开发时间线。

2015年7月–2019年8月,Hugo Penedone在谷歌DeepMind工作,从事深度学习与强化学习的应用研究
据他回忆,大约在2016年3月,DeepMind启动了AlphaFold 1,起因是在一次内部黑客马拉松Hackathon上,尝试将深度强化学习和优化算法应用于FoldIt游戏。
之后的几个月里,他们开始探索接触图(contact map)预测的可能性。

来自副溶血性弧菌(Vibrio parahaemolyticus)的蛋白质VPA0982的蛋白质接触图
由于在早期文献中,接触图已有相关概念,他们意识到,相较于直接预测整个蛋白质结构,使用神经网络来预测接触图的准确率更高。
因此,他认为DeepMind可能在2016年也独立地提出了这个不错的思路。
DeepMind的论文发布远晚于2016年NeurIPS的相关研究,显然,他们应该在工作中引用这些前人的成果!

针对此事,AI界当代最著名巨擘之一、Meta AI实验室灵魂人物Yann LeCun也表达了看法。
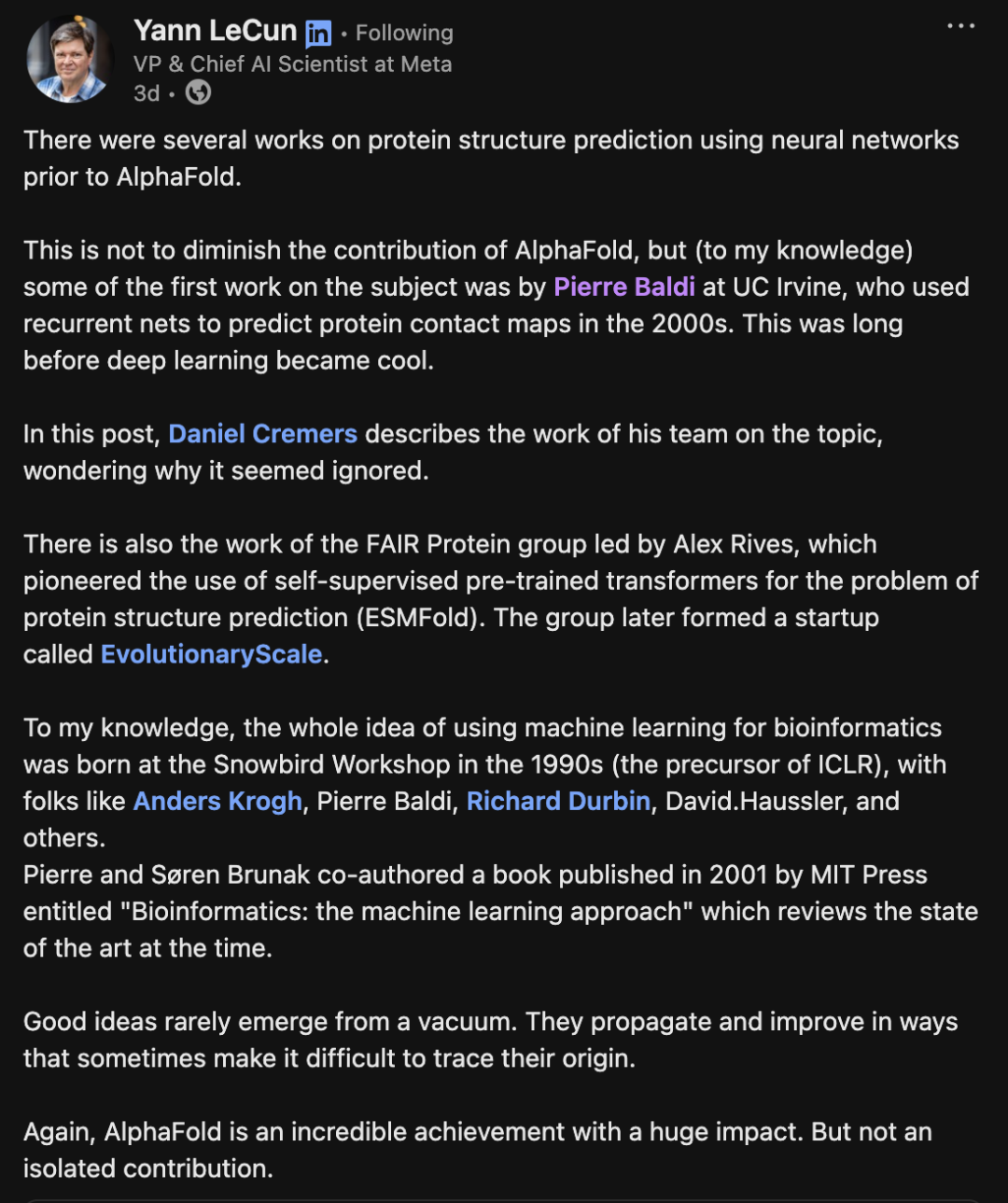
LeCun提到,1990年代的雪鸟研讨会(Snowbird Workshop)(ICLR的前身),使用机器学习进行生物信息学研究的整个想法就已诞生。
其中,与会者包括Anders Krogh(哥本哈根大学教授)、Pierre Baldi(加州大学欧文分校教授)、Richard Durbin(剑桥大学遗传学教授)、David Haussler(加州大学圣克鲁兹分校基因组学研究所科学总监)等人。



在AlphaFold之前,已有若干使用神经网络进行蛋白质结构预测的研究工作。
LeCun直言,没有贬低「AlphaFold贡献」的意思。
值得注意的是,最早在这个领域开展工作的是1990年代雪鸟研讨会与会者之一的加州大学欧文分校的Pierre Baldi。

他在2000年使用循环网络预测蛋白质接触图。

论文地址:https://pubmed.ncbi.nlm.nih.gov/11120677/
论文地址:https://pubmed.ncbi.nlm.nih.gov/10871264/
论文地址:https://pubmed.ncbi.nlm.nih.gov/10869034/
远早于深度学习变得流行之前。
LeCun一句话,引人深思:
好的想法很少凭空出现。它们以某种方式传播和改进,有时甚至难以追溯其起源。
LeCun直言,同样,AlphaFold是一项具有巨大影响力的非凡成就,但并非孤立的贡献。
最早在这个领域开展工作的加州大学欧文分校教授Pierre Baldi也表达了看法。
Pierre Baldi谈到,深度学习在某种蛋白质结构预测中的首次应用是在20世纪80年代。
当时,是由Qian和Sejnowski针对更简单的蛋白质二级结构预测问题开展的工作。

论文地址:https://pubmed.ncbi.nlm.nih.gov/3172241/
这样来看,用于预测接触图和蛋白质结构的深度学习方法,确实比AlphaFold早了二十年。
梳理来看,仔细查阅文献就会发现,用于预测接触图的深度学习方法,对图神经网络的发展也起到了重要作用。
「早在DeepMind之前,这些方法还被用于学习如何下围棋,而这一点DeepMind从未承认过。」Baldi指出。
Pierre Baldi直言不讳,「从长远来看,科学关乎真理与美。而在短期内,它是一项相当肮脏的人类事务。」
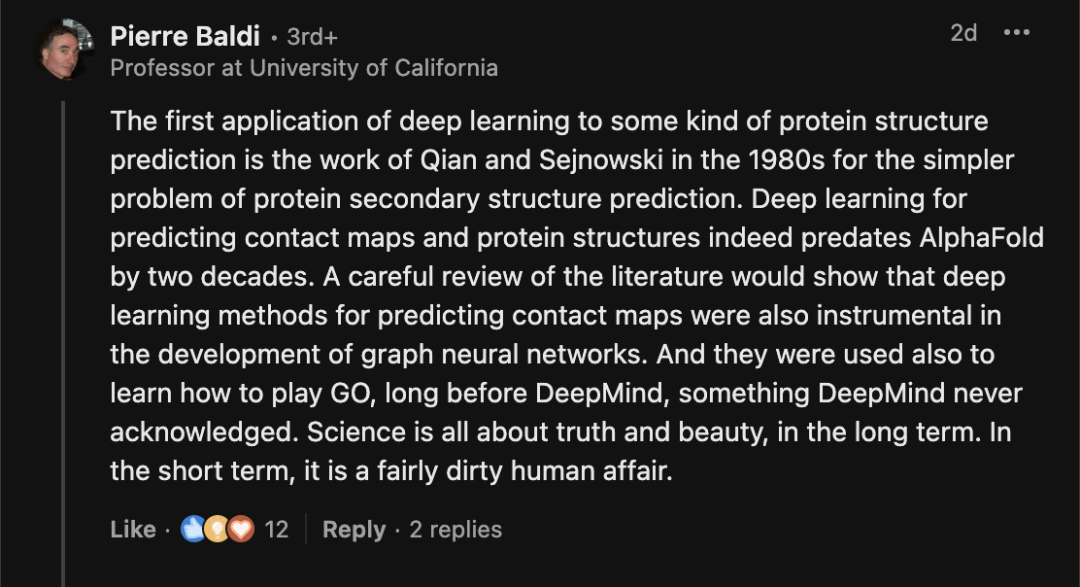
LeCun补充说道,「好的想法很少凭空出现。它们以某种方式传播和改进,有时甚至难以追溯其起源。」
同样,AlphaFold是一项具有巨大影响力的非凡成就,但并非孤立的贡献。
AlphaFold的成功固然值得赞誉,但正本清源,厘清其技术源流,更有助于理解AI在生命科学的深层逻辑和未来潜力。
当好的想法和研究推动社会发展时,希望更多人也能多关注核心研究背后的故事与核心人物。
正是他们的默默付出与耕耘,才有了能与大家见面的「奇迹」的诞生,推动社会向好发展。


 扫码添加微信
扫码添加微信
- 点赞 0
-
分享
微信扫一扫
-
加入群聊
 扫码加入群聊
扫码加入群聊